Využití programu ImageJ v rostlinné histologii
ImageJ in the practice of plant histology Kateřina Eliášová Mon, 05/18/2020 - 15:22Kateřina Eliášová & Libor Fettr
Úvod
Úvod Kateřina Eliášová Wed, 04/29/2020 - 12:04V rámci projektu MŠMT Inter-COST Aktivity NEUBIAS (a v rámci tréninkové školy analýzy obrazu, organizované během akce COST NEUBIAS), jsme vytvořili postup hodnocení histochemického průkazu obsahu fenolických látek v histologických řezech rostlinným materiálem. K digitálnímu zpracování mikroskopických snímků a kvantifikaci histochemického barvení jsme využili volně dostupný program ImageJ (https://imagej.net). Tato webová stránka by měla sloužit mikroskopikům - rostlinným anatomům, kteří řeší podobnou problematiku a pokoušejí se o kvantitativní analýzu svých mikroskopických snímků. Zájemci mohou celý postup sledovat na našich snímcích, případně pro své snímky využít námi vygenerovaná makra i model klasifikátoru, které jsou přiloženy jako přílohy ke stažení v příslušných kapitolách a lekcích nebo shlédnout instruktážní videa. Budeme také rádi, když experti v oboru analýzy obrazu pomocí ImageJ navštíví tuto webovou stránku a pomohou postup analýzy obrazu i podrobný návod vylepšit.
Postup ukážeme na konkrétním příkladu kvantifikace fenolických látek ve zralých somatických embryích smrku, který jsme využili v jednom z našich projektů. Na histologických řezech jsme studovali vliv ozáření UV-B na akumulaci fenolických látek v pletivech celých embryí (http://lbac.ueb.cas.cz/index.php/publikace/histological-and-biochemical…). Výsledky byly prezentovány formou posteru na 3. výroční konferenci NEUBIAS věnované analýze obrazu v biologii (https://lbac.ueb.cas.cz/publikace/quantification-histochemical-detection…). Domníváme se, že postup je dobře aplikovatelný i na hodnocení jiných barevných histochemických průkazů na řezech jiných rostlinných objektů.
Analýza obrazu
Analýza obrazu Kateřina Eliášová Wed, 04/15/2020 - 12:49Způsobů hodnocení histologických řezů je jistě mnoho. Předkládáme jednu z možností kvantifikace histochemického průkazu na řezech, který je založen na detekci rozdílů v barvení pletiv, buněk nebo buněčného obsahu. V těchto několika odstavcích stručně nastiňujeme postup hodnocení mikroskopických snímků řezů, který je rozveden do detailů v následujících kapitolách a lekcích.
Výchozím materiálem pro analýzu obrazu obecně jsou kvalitní snímky kvalitních, dobře obarvených histologických řezů. V našem případě to byly 5 µm silné řezy somatických embryí smrku zalitých do metakrylátové pryskyřice (Technovit 7100) a obarvených toluidinovou modří. Toto metachromatické barvivo barví rostlinné struktury různými barevnými odstíny podle jejich chemického složení. V případě řezů somatickými embryi smrku se barví modře celulózní buněčné stěny a jádra, zeleně nebo zeleno-modře fenolické látky uložené ve vakuolách buněk. Mikroskopické snímky vybraných řezů jsme analyzovali na základě těchto barevných rozdílů. Naším cílem bylo zjistit poměr plochy, kterou zaujímají buňky s obsahem fenolických látek ve vakuolách, vůči celé ploše řezu.
Pro hodnocení snímků jsme využili nástroje programu ImageJ (Rueden et al., 2017; Schindelin et al., 2012; Schindelin, J., et al., 2015; Schneider et al., 2012), který je volně ke stažení na stránce https://imagej.net nebo https://fiji.sc/. Mikroskopické snímky získané pomocí programu NIS-Elements firmy Laboratory Imaging a uložené ve formátu JPEG 2000 jsme museli nejprve v ImageJ upravit, abychom usnadnili analýzu obrazu.
Dalším postupným cílem bylo takové digitální zpracování obrazu, které by vedlo k odlišení buněk akumulujících fenolické látky od těch, které je neobsahují. To znamená, že bylo třeba provést tzv. segmentaci obrazu. Segmentace obrazu je soubor různých metod, které (slovy definice ve Wikipedii) "slouží k automatickému rozdělení vlastního obrazu na oblasti se společnými vlastnostmi, které obvykle mají nějaký smysluplný význam." Identifikace oblastí nebo struktur zájmu ve snímku umožňuje jejich kvantifikaci a další analýzu. K segmentaci snímků jsme využili plugin Trainable Weka Segmentation (TWS; Arganda-Carreras et al., 2017), který je integrován do ImageJ. Informace o tomto pluginu jsou k dispozici na stránce https://imagej.net/Trainable_Weka_Segmentation#Train_classifier. TWS umožňuje uživateli definovat třídy objektů nebo struktur na snímku. Tato metoda segmentace je založena na strojovém učení tzv. klasifikátoru, který se učí rozpoznávat struktury na snímku na základě barevných značek (traces), které do snímku ručně kreslí uživatel a ručně přiřazuje do předem nadefinovaných tříd jednotlivých struktur. Každé třídě je defaultně přiřazena jedna barva. Každá značka je tak vlastně jednou oblastí zájmu (region of interest - ROI). Podstatou této metody je klasifikace vlastností pixelů jednotlivých oblastí snímku přiřazovaných do tříd. Po tréninku je klasifikátor aplikovatelný na další obrázky, na kterých provede segmentaci.
Po několika kolech tréninku klasifikátoru a přidávání dalších značek do tříd jsme získali segmentovaný obraz se strukturami vyznačenými v barvách odpovídajících jednotlivým třídám a černobílé pravděpodobnostní mapy pro jednotlivé třídy. Tyto mapy ukazují všechny pixely, které s největší pravděpodobností náležejí do dané třídy. V našem případě jsme nadefinovali čtyři třídy - pozadí (background), buněčná stěna (cell wall), buňky bez fenolických látek (cell no phenolics) a buňky s fenolickými látkami (cell with phenolics). Získali jsme proto jeden barevný segmentovaný obraz (označen v TWS jako Classified image) a čtyři pravděpodobnostní mapy.
Pravděpodobnostní mapu třídy buňky s fenoly jsme pomocí nástrojů programu ImageJ naprahovali tak, abychom vytvořili dvě oblasti zájmu - ROI. Jednu pro celý řez a druhou pro oblast buněk obsahujících fenolické látky. Plochu těchto dvou ROI jsme změřili.
Výsledkem analýzy obrazu byl odhad plochy, kterou zaujímají buňky akumulující fenoly, v ploše celého řezu. Přesnost tohoto odhadu závisí na úspěšnosti segmentace obrazu.
Příprava obrázku před tréninkem klasifikátoru
Příprava obrázku před tréninkem klasifikátoru Kateřina Eliášová Wed, 04/15/2020 - 22:56V ImageJ je možné otevřít a analyzovat snímky v nejrůznějších formátech. Obecně doporučen jako vhodný je formát TIFF, naopak jako nevhodný pro analýzu (protože při ukládání do tohoto formátu dochází ke ztrátě dat) je JPEG. Většina mikroskopiků nicméně získává snímky primárně ve formátu, do kterého ukládají snímky ovládací programy jejich mikroskopů. Tyto tzv. bio-formáty (např. ZEISS -.czi a .lsm; Laboratory imaging - .lim; NIS-Elements- .jp2 a .nd2) v ImageJ bez problémů otevřete a zanalyzujete.
My jsme snímali obrázky pomocí programu NIS-Elements ve formátu JPEG 2000, tedy s koncovkou .jp2, ve vysokém rozlišení. Abychom mohli snímky analyzovat, bylo třeba je nejprve upravit. Tyto úpravy ukazujeme v této kapitole v pěti podřízených lekcích.
Pokud vaše výchozí originální snímky budou uloženy v jiném formátu s jiným rozlišením apod., nebude možná nutné snímky upravovat. Je třeba to vyzkoušet. Obecně platí, že příliš velké snímky s velkým rozlišením se touto metodou obtížně analyzují a segmentace obrazu vůbec nepůjde nebo bude trvat neúměrně dlouho. Celý proces také závisí na vlastnostech počítače.
Proces úpravy snímku jsme zaznamenali v záznamníku ImageJ (Plugins>Macro>Record) a vytvořené Makro přikládáme. Můžete si ho stáhnout a spustit v ImageJ v nabídce Plugins>New>Macro vložením a "Run".
// Makro pro přípravu souboru JP2 před tréninkem.
//
// Uživatel vybere soubor, pravděpodobně s příponou JP2.
path = File.openDialog("Select a JP2 File");
// Otevře soubor.
open(path);
// Připraví název výsledného souboru.
orig_image_title = getTitle();
dotIndex = indexOf(path, ".");
results_name = substring(path, 0, dotIndex) + " (prepared)";
// Připraví soubor.
lbac_prepare_image(path, results_name);
CloseAllWindows();
message_title = "Obrázek je připraven pro trénink";
message = "Obrázek '" + orig_image_title + "' je připraven pro trénink pod jménem '" + results_name + ".tif'";
showMessage(message_title, message);
/**
*Funkce pro přípravu obrázku.
*/
function lbac_prepare_image(path, new_image_title) {
// Převede do RGB.
run("RGB Color");
// Upraví měřítko.
run("Scale...", "x=0.5 y=0.5 width=1280 height=960 interpolation=Bilinear average create");
// Upraví kontrast a LUT (look-up table).
//
run("Brightness/Contrast...");
title = "Adjust Brightness/Contrast";
msg = "V dialogu B&C upravte nastavení jasu a kontrastu a potvrďte 'Apply'.\nPro pokračování klepněte na 'OK'.";
waitForUser(title, msg);
getMinAndMax(min,max); // Returns the minimum and maximum displayed pixel values (display range).
if (min != 0 && max != 255) { // Check if the display has been changed.
run("Apply LUT");
}
selectWindow("B&C");
run("Close");
// Přejmenuje a uloží VÝSLEDNÝ soubor ve formátu tif.
saveAs("tif", new_image_title);
}
// "Close All Image Windows"
function CloseAllWindows() {
while (nImages>0) {
selectImage(nImages);
close();
}
}
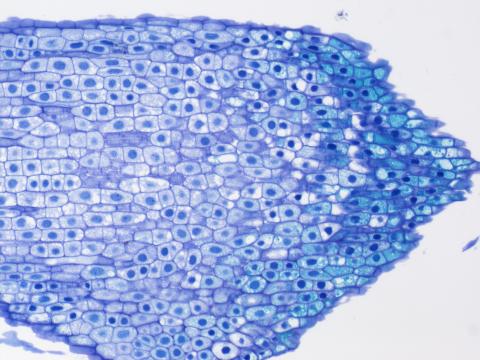
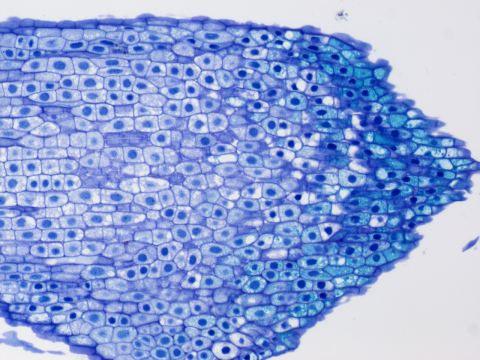
1 – Výchozí obrázek
1 – Výchozí obrázek1 – výchozí originální snímek, pro potřeby webové prezentace uložený jako JPEG, je pouze ilustrační. V ImageJ jsme otevřeli originální snímek v tzv. bio-formátu programu NIS-Elements. V tomto případě to je JPEG 2000 s koncovkou .jp2. V tomto formátu je snímek ke stažení v příloze.
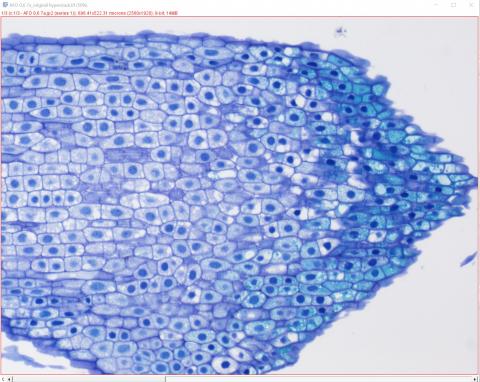
2 - Otevření snímku v ImageJ
2 - Otevření snímku v ImageJ2 – Výchozí originální snímek jsme otevřeli v ImageJ (File>Open nebo přetažením na lištu (Status Bar) - Drag&Drop) jako Hyperstack bez zaškrtnutého políčka Autoscale. Vyzkoušejte si, jak váš snímek vypadá po otevření bez nebo s Autoscale. Tato funkce upravuje minimální a maximální hodnoty histogramu a může změnit barevné odstíny v obrázku. Pokud při histochemickém barvení opravdu záleží na odstínech jako v případě detekce fenolických látek pomocí barvení toluidinovou modří, je třeba dát pozor. Kontrast můžete později upravit jinak (Image>Adjust>Brightness&Contrast).
V záhlaví snímku jsou vždy kromě názvu souboru uvedeny informace o typu a velikosti snímku v mikrometrech, pixelech, bitech i bytech.
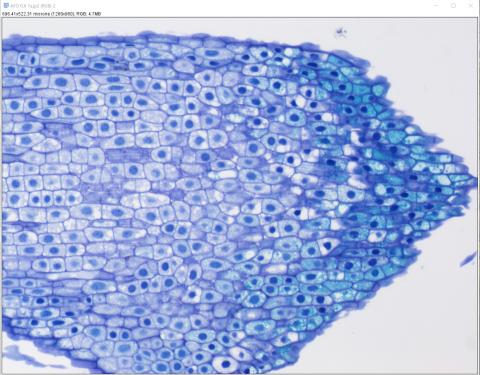
3 - Převedení snímku do RGB
3 - Převedení snímku do RGB3 – Snímek jsme otevřeli jako Hyperstack, proto je programem považován za vícekanálový. Abychom s ním mohli dál pracovat, bylo třeba převést snímek na prostý RGB – (Image>Type>RGB color).
4 - Zmenšené rozlišení
4 - Zmenšené rozlišení4 – RGB obrázek má velké rozlišení (2560x1920 pixelů). Abychom usnadnili segmentaci obrazu, počet pixelů jsme zmenšili na polovinu (Image>Scale - X scale 0.5, Y Scale 0.5; Average when downsizing, Create new window).
5 - Snímek s upraveným kontrastem
5 - Snímek s upraveným kontrastem5 – Snímek jsme otevřeli bez Autoscale, proto jsme upravili kontrast (Image>Adjust> Brightness & Contrast – Auto-Apply). Pokud je výsledek úpravy kontrastu pomocí tlačítka Auto neuspokojivý (snímek je např. příliš tmavý), je možné kontrast upravit pomocí nastavení min./max. hodnot histogramu, LUT – nastavujte stejně všechny obrázky, které budete analyzovat.
Trénink klasifikátoru
Trénink klasifikátoru Kateřina Eliášová Wed, 04/15/2020 - 21:44Upravený snímek (případně originál, který úpravu nevyžadoval) vstupuje do tréninku klasifikátoru. Zvolíme plugin TWS (Plugins>Segmentation>Trainable Weka Segmentation) – snímek se otevře znovu v klasifikátoru. Pak nastupuje ruční označování struktur značkami (traces) a jejich přiřazování do tříd, trénink klasifikátoru a další úprava značek, přidání dalších tříd, dokud výsledná segmentace neodpovídá strukturám, které hodnotíme. Výsledný klasifikátor uložíme spolu s datovým souborem, který obsahuje informace o klasifikaci pixelů (ve formě vektorů), náležejících jednotlivým značkám. Výsledkem "práce" klasifikátoru je barevný segmentovaný obraz (Classified image) a pravděpodobnostní mapy (Probability maps) jednotlivých tříd, do kterých jsme přiřadili značky pro jednotlivé struktury. Segmentovaný obraz je výsledkem mnoha matematických operací, které probíhají na pozadí při tréninku klasifikátoru nebo při generování výsledků. Doba, po kterou tyto operace probíhají, závisí na vlastnostech počítače. Postup jsme po jednotlivých krocích popsali v následujících lekcích.
Několik úvodních kroků spuštění klasifikátoru je v přiloženém makru.
// Makro pro spuštění tréninku klasifikátoru Weka.
// Viz https://imagej.net/Trainable_Weka_Segmentation#Complete_macro_example:
// Uživatel vybere soubor, pravděpodobně s příponou TIF.
path = File.openDialog("Select a TIF File");
delay = 1000;
twsWindowName = "Trainable Weka Segmentation v3.2.34";
// Pokud nechcete pomocné texty, nastavte "false":
showMessages = true;
open(path);
// Spustíme TWS.
run("Trainable Weka Segmentation");
// Lepší je počkat.
wait(delay);
selectWindow(twsWindowName);
// Připravné operace.
call("trainableSegmentation.Weka_Segmentation.setFeature", "Hessian=false");
call("trainableSegmentation.Weka_Segmentation.setMembranePatchSize", "16");
call("trainableSegmentation.Weka_Segmentation.setMaximumSigma", "8.0");
// Přejmenování a přidání tříd.
call("trainableSegmentation.Weka_Segmentation.changeClassName", "0", "background");
call("trainableSegmentation.Weka_Segmentation.changeClassName", "1", "cell wall");
call("trainableSegmentation.Weka_Segmentation.createNewClass", "cell no phenolics");
call("trainableSegmentation.Weka_Segmentation.createNewClass", "cell with phenolics");
// Stanovení "oblasti zájmu" třídy - kde bude probíhat trénink pro danou třídu.
// Možná by se oblast zájmu mohla nastavit i pro ostatní třídy?
/*
makeRectangle(0, 0, 100, 100);
title = "Vyberte ROI pro cell wall";
msg = "Vyberte obdélník\nPro pokračování klepněte na 'OK'.";
waitForUser(title, msg);
// add one region of interest to each class
call("trainableSegmentation.Weka_Segmentation.addTrace", "1", "1");
*/
// BACKGROUND
run("Line Width...", "line=50");
setTool("freeline");
wait(delay);
if (showMessages) {
msg = "<html>Vyberte vzorky pro trénink pozadí a přidávejte je tlačítkem <strong>Add to background</strong>.<br />"
+ "Šířku linky upravte poklepáním na Freehand lines.</html>";
showMessage(msg);
}
1 - Otevření upraveného obrázku v pluginu Trainable Weka Segmentation
1 - Otevření upraveného obrázku v pluginu Trainable Weka SegmentationV nabídce ImageJ zvolíme plugin TWS (Plugins>Segmentation>Trainable Weka Segmentation) – otevřený snímek se otevře znovu v okně klasifikátoru. Pokud není žádný snímek otevřený, otevře se složka, ve které jsou připravené snímky uložené s výzvou k otevření snímku.
Na tréninkovém panelu vlevo (Training) se nacházejí tlačítka pro trénink klasifikátoru a generování výsledků: Train classifier spouští tréninkový proces; Toggle overlay umožňuje překlikávat mezi obrázkem s nakreslenými značkami a překryvným barevným výsledkem tréninku klasifikátoru a porovnávat jak se dosažená segmentace shoduje se strukturami na snímku; Create results generuje a ukazuje výsledný barevný segmentovaný obraz; Get probability na základě aktuálního klasifikátoru zobrazuje pravděpodobnost, že každý pixel patří do definované třidy ve formě pravděpodobnostních map.
Pod tréninkovým panelem je panel možností (Options): Apply classifier umožňuje aplikovat aktuální klasifikátor na jakýkoli snímek, připravený v souboru pro segmentaci (je třeba otevřít snímek a zvolit zda výsledkem má být segmentovaný obraz nebo pravděpodobnostní mapa); Load classifier umožňuje načíst dříve vytvořený a uložený klasifikátor a aplikovat na aktuální snímek; Save classifier uloží aktuální klasifikátor ve formátu .model pro pozdější použití. Load data načte data z předcházejících značek nakreslených do aktuálního nebo jiného snímku. Save data uloží data o aktuálně vytvořených značkách ve formátu ARFF. Create new class umožní vytvořit a pojmenovat novou třídu; výchozí počet tříd, se kterým klasifikace začíná, jsou dvě. V Settings je možné měnit matematické parametry klasifikace pixelů, přejmenovat třídy nebo změnit průhlednost překryvného výsledného obrazu, aby pod ním byl nebo nebyl vidět původní snímek histologického řezu (Results overlay opacity - výchozí nastavení je na hodnotě 33).
Na pravé straně se nachází panel se seznamem značek pro každou třídu a tlačítka pro přidání značky (tj. aktuálního ROI) do specifické třídy.
2 - Přiřazení struktur do dvou tříd a první běh tréninku klasifikátoru
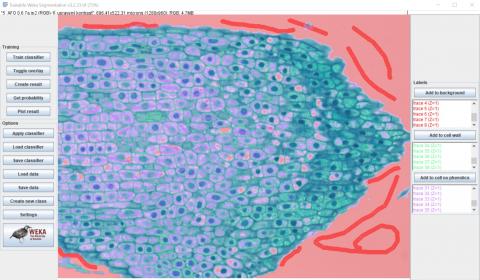
2 - Přiřazení struktur do dvou tříd a první běh tréninku klasifikátoru Kateřina Eliášová Thu, 04/16/2020 - 23:43V předchozím textu jsme zmínili, že jsme pro segmentaci obrazu zvolili čtyři třídy. Podle výchozího nastavení ovšem začíná plugin TWS se dvěma třídami. Pokud histologický řez nevyplňuje celý snímek, je na počátku tréninku klasifikátoru vhodné odlišit vlastní řez od okolního pozadí. Proto jsme nejprve definovali třídy pozadí (background) a buněčná stěna (cell wall). Další dvě třídy jsme přidávali postupně, vždy po tréninku klasifikátoru.
Značky do tříd je možné přiřazovat pomocí nástrojů pro kreslení ROI, které jsou k dispozici v nástrojovém menu ImageJ - použít lze obdélník, ovál, elipsu, polygon, štětec nebo od ruky kreslenou linku (freehand line), která má ve výchozím nastavení šířku 1 pixelu. Linka od ruky je automaticky vybrána při spuštění TWS a i my jsme ji používali výhradně. Šířku linky můžeme změnit v okně, které se otevře po dvojkliknutí levým tlačítkem myši na ikonu linky od ruky. Širší linku jsme používali pro značení celých buněk a pro označení pozadí kolem řezu, pro značení buněčných stěn byla vhodnější linka tenčí. Snímek můžeme zvětšovat nebo zmenšovat tlačítky "+" a "-" nebo po zapnutí lupy na nástrojové liště ImageJ také levým a pravým tlačítkem myši.
Každou značku je třeba ručně přiřadit do třídy hned po nakreslení. Nepřiřazená značka po nakreslení další značky zmizí. Značku, kterou jsme po nakreslení přiřadili do třídy, ale nejsme spokojeni, s tím, jak jsme ji nakreslili nebo jsme se spletli a zařadili ji do špatné třídy, zrušíme dvojklikem levou klapkou myši na zápis v seznamu na pravé straně okna TWS. Není možné značku smazat přímo v obrázku. Po smazání v seznamu změní značka na obrázku barvu a po kliknutí na jiné místo ve snímku zmizí.
Na horním obrázku ukazujeme první nakreslené a do seznamů dvou tříd přidané značky.
Na dolním obrázku ukazujeme výsledek prvního kola tréninku klasifikátoru. Je zřejmé, že klasifikátor pouze odlišil řez od pozadí, do kterého zařadil i některé velké vakuoly.
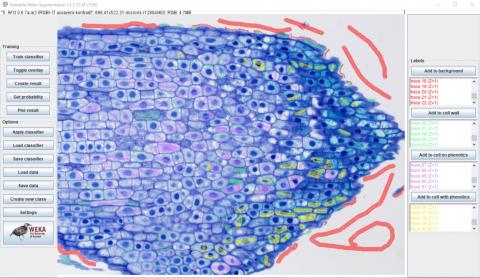
3 - Přidání další klasifikační třídy, označení struktur, další běh tréninku
3 - Přidání další klasifikační třídy, označení struktur, další běh tréninku Kateřina Eliášová Thu, 04/16/2020 - 23:49Dalším krokem po proběhnutí prvního tréninku klasifikátoru bylo nadefinování další třídy - buňky bez fenolů (Create new class) (viz horní obrázek). Značky, kterými jsme chtěli tyto ROI přiřadit do třídy, jsme kreslili přes celé buňky, snažili jsme se zahrnout celý obsah buněk včetně jader a vakuol. Samostatnou třídu buněčná jádra jsme nedefinovali, protože naším cílem bylo rozlišit celé buňky podle obsahu vakuol (+fenoly x -fenoly) a ne podle velikosti nebo tvaru jádra. Po přidání značek i do ostatních tříd, především do třídy buněčná stěna, jsme znovu spustili trénink klasifikátoru. Výsledek ukazuje prostřední obrázek. Je zřejmé, že se už do značné míry podařila segmentace buněčných stěn, ale stále ještě jsou některé vakuoly přiřazovány do třídy pozadí (překryvná barva je červená). Výsledek dalšího přidání značek do chybně klasifikovaných míst a následného tréninku ukazuje spodní obrázek. Je vidět, že stále méně vakuol je přiřazováno chybně, ale jádra jsou vzhledem k výraznému zbarvení často přiřazována do třídy buněčná stěna (překryvná barva je zelená).
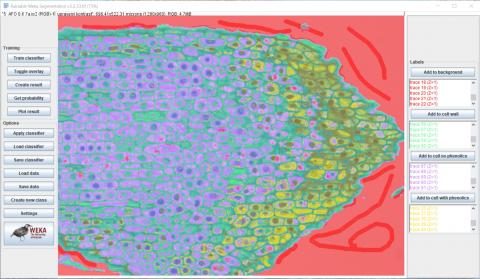
4 - Přidání čtvrté klasifikační třídy a čtvrtý běh tréninku
4 - Přidání čtvrté klasifikační třídy a čtvrtý běh tréninku Kateřina Eliášová Thu, 04/16/2020 - 23:53Dalším krokem bylo vytvoření poslední, čtvrté třídy - buňky s fenoly (cell with phenolics). Na horním obrázku jsou vidět do snímku přidané žluté značky. Na spodním obrázku je výsledek následného tréninku klasifikátoru se žlutě vyznačenou oblastí buněk s obsahem fenolických látek především na pravé straně snímku.
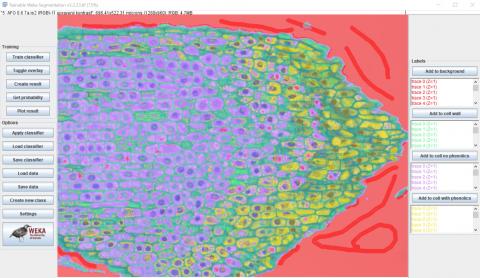
5 - Přidání značek a poslední běh tréninku
5 - Přidání značek a poslední běh tréninku Kateřina Eliášová Fri, 04/17/2020 - 00:18Dalším přidáním značek a jejich přiřazením do příslušných tříd (viz horní obrázek) jsme se snažili zpřesnit segmentaci snímku (viz dolní obrázek). Vzhledem k uspokojivým výsledkům jsme se rozhodli ukončit trénink klasifikátoru. Výsledný model klasifikátoru a příslušná data o klasifikaci pixelů v jednotlivých ROI jsme uložili. Obojí je ke stažení v příloze.
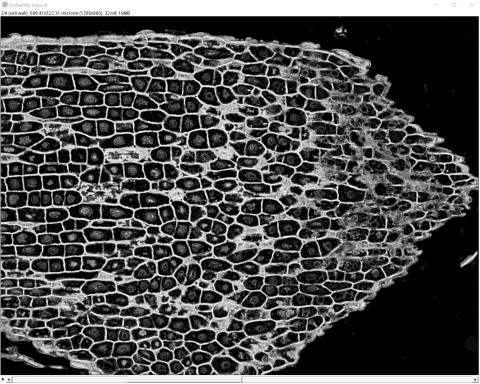
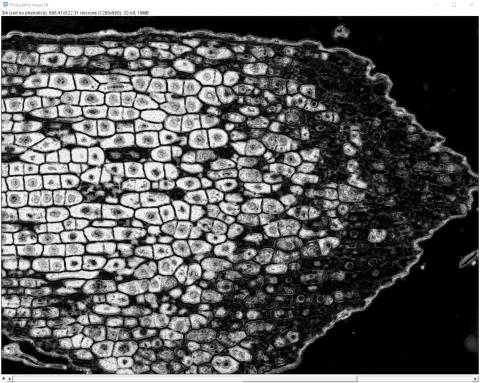
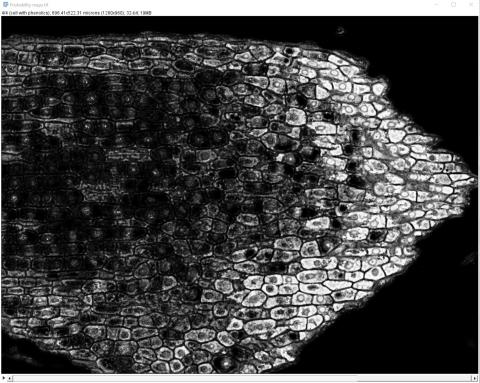
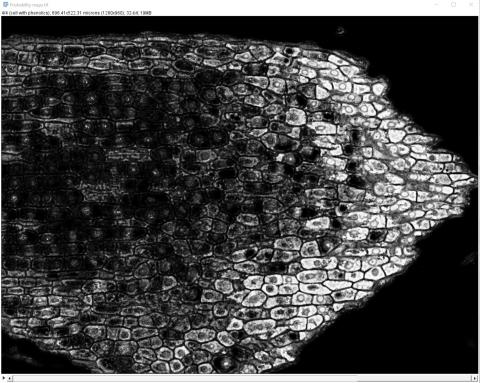
6 - Výsledný segmentovaný obrázek a pravděpodobnostní mapy
6 - Výsledný segmentovaný obrázek a pravděpodobnostní mapy Kateřina Eliášová Fri, 04/17/2020 - 00:21Po ukončení tréninku klasifikátoru jsme vygenerovali (Create results) výsledný segmentovaný obraz (Classified image - horní barevný obrázek) a čtyři pravděpodobnostní mapy (Probability maps - černobílé obrázky pro jednotlivé třídy v pořadí od shora - pozadí, buněčná stěna, buňky bez fenolů, buňky s fenoly). Pravděpodobnostní mapa třídy buňky s fenoly se stala výchozím snímkem pro další kvantitativní analýzu, kterou popisujeme v kapitole Měření.
Na segmentovaném snímku jsou oblasti spadající do jednotlivých tříd vyznačeny příslušnými barvami. Je zřejmé, že segmentace nebyla dokonalá, protože v buňkách bez obsahu fenolických látek (zbarveny fialově) jsou některá jádra částečně přiřazena do zelené třídy buněčná stěna a vyskytují se zde malé oblasti přiřazené do třídy buňky s fenoly; některé části vakuol v obou typech buněk byly přiřazeny do červené třídy pozadí. To znamená, že jsme trénink klasifikátoru ukončili příliš brzy a bylo třeba ještě pokračovat nebo je segmentace příliš obtížná vzhledem k podobnosti v odstínech barvení jader a buněčných stěn. Případně očima nevidíme zelenavé zbarvení v některých buňkách, které klasifikátor odhalil.
Na černobílých snímcích pravděpodobnostních map je daná třída vždy vykreslena bíle.
Použití klasifikátoru na více obrázků
Použití klasifikátoru na více obrázků Kateřina Eliášová Tue, 04/28/2020 - 09:10Vytvořený klasifikátor lze pomocí skriptu v ImageJ použít automaticky na více souborů. Příklad takového skriptu je níže. Pro skriptování pluginu Trainable Weka Segmentation (TWS) lze používat standardní makro jazyk ImageJ. Často se ale dává přednost skriptovacímu jazyku Bean Shell. Tak je tomu i v tomto případě.
Skript pro použití klasifikátoru na více souborů jsme převzali z příručky pro skriptování TWS. Můžete ho spustit v ImageJ (Plugins -> New -> Macro) po vložení do skriptovacího formuláře a klepnutím na "Run". Před spuštěním změňte skriptovací jazyk z IJ1 Macro na Bean Shell v nabídce Language formuláře.
V makru po spuštění zadáme
- vstupní složku s připravenými soubory tif
- výstupní složku (jiná než vstupní)
- soubor klasifikátoru (.model)
- režim výsledku
Další běh makra už je bez interakce s uživatelem.
#@ File(label="Input directory", description="Select the directory with input images", style="directory") inputDir
#@ File(label="Output directory", description="Select the output directory", style="directory") outputDir
#@ File(label="Weka model", description="Select the Weka model to apply") modelPath
#@ String(label="Result mode",choices={"Labels","Probabilities"}) resultMode
import trainableSegmentation.WekaSegmentation;
import trainableSegmentation.utils.Utils;
import ij.io.FileSaver;
import ij.IJ;
import ij.ImagePlus;
// starting time
startTime = System.currentTimeMillis();
// caculate probabilities?
getProbs = resultMode.equals( "Probabilities" );
// create segmentator
segmentator = new WekaSegmentation();
// load classifier
segmentator.loadClassifier( modelPath.getCanonicalPath() );
// get list of input images
listOfFiles = inputDir.listFiles();
for ( i = 0; i < listOfFiles.length; i++ )
{
// process only files (do not go into sub-folders)
if( listOfFiles[ i ].isFile() )
{
// try to read file as image
image = IJ.openImage( listOfFiles[i].getCanonicalPath() );
if( image != null )
{
// apply classifier and get results (0 indicates number of threads is auto-detected)
result = segmentator.applyClassifier( image, 0, getProbs );
if( !getProbs )
// assign same LUT as in GUI
result.setLut( Utils.getGoldenAngleLUT() );
// save result as TIFF in output folder
outputFileName = listOfFiles[ i ].getName().replaceFirst("[.][^.]+$", "") + " out.tif";
new FileSaver( result ).saveAsTiff( outputDir.getPath() + File.separator + outputFileName );
// force garbage collection (important for large images)
result = null;
image = null;
System.gc();
}
}
}
// print elapsed time
estimatedTime = System.currentTimeMillis() - startTime;
IJ.log( "** Finished processing folder in " + estimatedTime + " ms **" );
Měření
Měření Kateřina Eliášová Thu, 04/16/2020 - 18:39V této kapitole ukážeme, jak můžeme výsledky segmentace obrazu využít ke kvantitativní analýze. Výchozí obrázek pro měření bude hyperstack čtyř pravděpodobnostních map, který byl výsledkem segmentace obrazu pomocí klasifikátoru.
Celý proces provede přiložené makro. Můžete ho spustit v ImageJ v nabídce Plugins>New>Macro vložením a "Run".
// Makro prahování, tvorba ROI, měření včetně otevření hyperstacku a duplikace mapy buněk s fenoly
// http://lbac.ueb.cas.cz/strana-knihy/mereni
//
// Uživatel vybere soubor, pravděpodobně s příponou TIF.
path = File.openDialog("Select probability maps TIF File");
cell_with_phenolics_image_name = "cell with phenolics";
Dialog.create("Confirm image name");
Dialog.addMessage("Prosím, potvrďte nebo upravte název obrázku pro měření.\n ");
Dialog.addString("Název obrázku:", cell_with_phenolics_image_name, 25);
Dialog.show();
cell_with_phenolics_image_name = Dialog.getString();
// Otevře soubor.
open(path);
// Připraví název výsledného souboru.
orig_image_title = getTitle();
dotIndex = indexOf(path, ".");
results_name = substring(path, 0, dotIndex) + " (measurement results)";
// Měří.
lbac_measurement_area(path, cell_with_phenolics_image_name, results_name);
message_title = "Výsledek měření byl uložen";
message = "Výsledek měření byl uložen do souboru " + results_name
+ "\nOkna s obrázky budou zavřena bez ukládání.";
showMessage(message_title, message);
while (nImages>0) {
selectImage(nImages);
close();
}
/**
* Změří a uloží výsledky do CSV.
*/
function lbac_measurement_area(path, image_name, results_file_title) {
run("Stack to Images");
selectWindow(image_name);
run("Duplicate...", " ");
setAutoThreshold("Default dark");
//run("Threshold...");
//setTool("wand");
doWand(1054, 318);
run("Create Selection");
// ROI - region of interest
run("ROI Manager...");
roiManager("Add");
roiManager("Select", 0);
roiManager("Rename", "phenolics");
selectWindow(image_name);
setAutoThreshold("Default dark");
//setTool("wand");
doWand(1094, 414);
roiManager("Add");
roiManager("Select", 1);
roiManager("Rename", "full");
roiManager("Deselect");
run("Set Measurements...", "area display redirect=None decimal=3");
roiManager("Measure");
saveAs("Results", results_file_title + ".csv");
}
1 - Vstupní obrázek a jeho kopie
1 - Vstupní obrázek a jeho kopieV ImageJ jsme otevřeli hyperstack s pravděpodobnostními mapami všech tříd, který jsme získali segmentací obrazu pomocí TWS. Rozdělili na jednotlivé mapy (Image>Stacks>Stack to Images) a vytvořili kopii pravděpodobnostní mapy "buňky s fenoly" (Image>Duplicate).
2 - Prahování a ROI oblasti buněk s fenoly
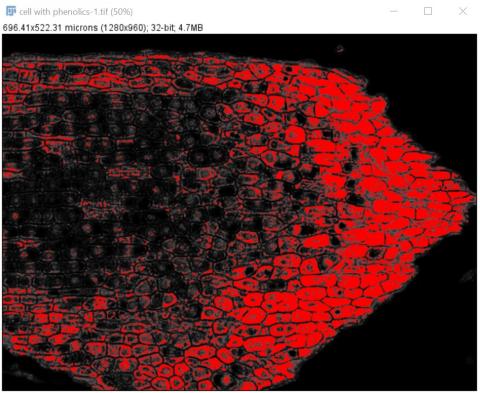
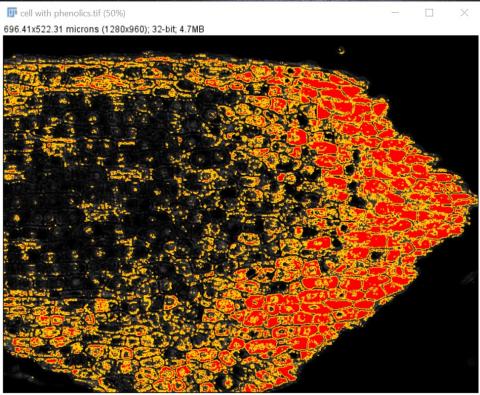
2 - Prahování a ROI oblasti buněk s fenoly Kateřina Eliášová Sun, 04/19/2020 - 23:38Původní pravděpodobnostní mapu buněk s fenoly jsme naprahovali červeně tak, abychom vyznačili oblast buněk s fenoly (Image>Adjust>Threshold AUTO). Do červené oblasti jsme klikli kouzelnou hůlkou (magic wand v nástrojovém menu ImageJ). Vytvořil se malý částečný ROI. Abychom získali ROI celé oblasti, nastavili jsme výběr (Edit>Selection>Create Selection). Otevřeli jsme ROI manager (Analyze>Tools>ROI manager) a přidali do něj vyznačenou oblast (Add, možno udělat i klávesovou zkratkou t), kterou jsme vzápětí přejmenovali (Rename) "phenolics" .
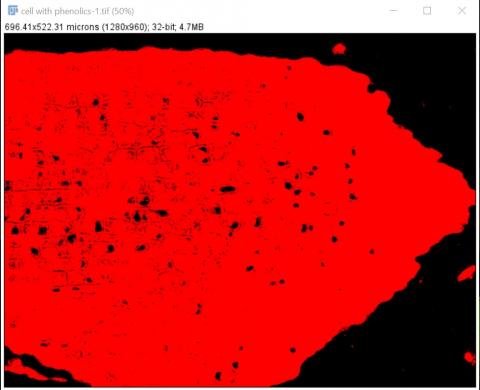
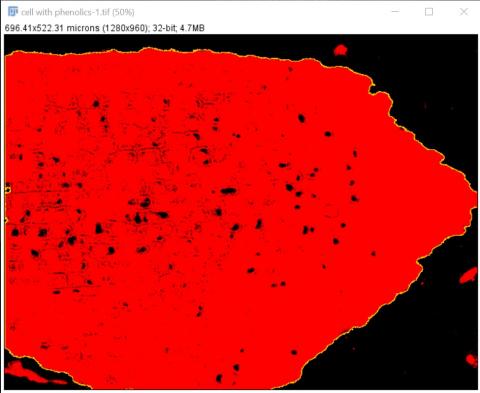
3 - Prahování a získání ROI celého řezu
3 - Prahování a získání ROI celého řezu Kateřina Eliášová Sun, 04/19/2020 - 23:25Zkopírovanou pravděpodobnostní mapu jsme naprahovali tak, aby byl vyznačený celý řez pletivem (Image>Adjust>Threshold; zaklikli jsme AUTO, čímž se vyznačila oblast s fenoly a pak jsme upravili práh tak, aby červená zakryla pokud možno celý řez). Poté jsme klikli kouzelnou hůlkou (magic wand v nástrojovém menu ImageJ) do červené oblasti - vyznačil se žlutě ROI kolem celého řezu. Otevřeli jsme ROI manager (Analyze>Tools>ROI manager) a přidali do něj vyznačenou oblast (Add, možno udělat i klávesovou zkratkou t), kterou jsme vzápětí přejmenovali (Rename) "Full", jako celý řez.
4 - Měření plochy ROI
4 - Měření plochy ROI Kateřina Eliášová Sun, 04/19/2020 - 23:48Nastavili jsme parametry měření (Analyze>Set measurements) - zaškrtli jsme AREA a DISPLAY LABELS. V ROI manageru jsme zvolili Deselect (abychom neměřili plochu jen v jednom zvoleném ROI, ale v obou) a následně Measure. Získali jsme tabulku se změřenými plochami obou ROI, kterou je možné uložit jako soubor Excel.
Celý postup prahování, vytvoření ROI a změření jejich plochy je ke shlédnutí v přiloženém videu.
Souhrn
Souhrn Kateřina Eliášová Wed, 04/15/2020 - 13:13Výběr řezů k hodnocení je zásadní z několika důvodů. Především kvalita řezů i barvení musí být vysoká, protože od ní se odvíjí možnosti a úspěšnost digitálního zpracování obrazu. Dále je třeba zvážit jaké řezy a kolik vybrat z hlediska morfologicko-anatomického uspořádání studovaného objektu, abychom získali nezatížený odhad parametrů, které kvantifikujeme. Je vhodné vzít v úvahu metody rovnoměrně náhodného výběru řezů, využít stereologické metody a tím snížit množství hodnocených řezů. My jsme brali v úvahu i to, kde se fenolické látky v embryu hromadí. Abychom mohli porovnávat ošetřené varianty s neošetřenými kontrolními vzorky, snažili jsme se vybírat řezy ze všech morfologických částí embrya (tj. děloh, hypokotylu a kořenové špičky s čepičkou) i anatomických struktur (pokožky, podpokožkového parenchymu i středního válce).
Rostlinné buňky mají mnoho nevýhod, které mikroskopikům komplikují život. V našem případě byla takovou komplikací přítomnost větších vakuol bez zjevného obsahu, která znesnadňovala segmentaci obrazu. Klasifikátor tyto zdánlivě prázdné prostory přiřazoval do třídy pozadí a dal se jen těžko přesvědčit, aby to nedělal. V takovém případě je nejspíš vhodné použít pro zakreslení značky jiný nástroj pro kreslení ROI než jen linku, byť o velké tloušťce. Použití polygonu nebo volného obrazce, který vykryje celou buňku, včetně jádra a vakuoly bez fenolických látek, může segmentaci usnadnit a zpřesnit. Výhodou pro kvalitní a správnou segmentaci je určitě kontrastní barvení různými barvivy. Stejný odstín pro buněčnou stěnu i jádro byl v našem případě problém.
Vytvořený model klasifikátoru je třeba vyzkoušet na dalších obdobných snímcích dřív, než ho aplikujeme na velký soubor obrázků. Doporučujeme trénink provést na více než jednom snímku, a zkontrolovat, zda klasifikátor přiřazuje struktury do tříd správně a výsledná segmentace obrazu odpovídá pravdě.
Poděkování
Poděkování Kateřina Eliášová Mon, 04/20/2020 - 17:25Práce byla podpořena grantem MŠMT č. LTC 17030. Metoda analýzy obrazu (úprava, segmentace a měření) histologického řezu byla vyvinuta v rámci tréninkové školy akce COST NEUBIAS, především díky laskavé pomoci Fabrice Cordelièresa (Bordeaux Imaging Center CNRS, Univerzita Bordeaux). Webová aplikace byla vytvořena ve spolupráci s naším web-masterem Liborem Fettrem.
Literatura
Literatura Kateřina Eliášová Mon, 04/20/2020 - 18:50Arganda-Carreras, I., Kaynig, V., Rueden, C., Eliceiri, K.W., Schindelin, J., Cardona, A., et al. (2017). Trainable Weka Segmentation: a machine learning tool for microscopy pixel classification. Bioinformatics 33(15), 2424-2426. doi: 10.1093/bioinformatics/btx180.
Schindelin, J., Arganda-Carreras, I., Frise, E., Kaynig, V., Longair, M., Pietzsch, T., et al. (2012). Fiji: an open-source platform for biological-image analysis. Nature Methods 9, 676. doi: 10.1038/nmeth.2019
Schindelin, J., Rueden, C. T., Hiner, M. C., Eliceiri, K. W. (2015). The ImageJ ecosystem: An open platform for biomedical image analysis. Molecular Reproduction and Development 82(7-8): 518-529. doi:10.1002/mrd.22489
Schneider, C. A., Rasband, W. S., Eliceiri, K. W. (2012). NIH Image to ImageJ: 25 years of image analysis. Nature Methods 9(7): 671-675. doi: 10.1038/nmeth.2089